推荐产品
公司新闻/正文
Science 新技术——细胞状态&分化命运图谱重构全景分化轨迹
人阅读 发布时间:2021-03-09 09:17

在成人体内造血干细胞(HSC)和祖细胞(progenitor)驻留在骨髓中维持稳态的造血功能,数十年的细胞培养和移植研究已构建出看似完整的造血祖细胞群的分化层级关系,但近期的 scSeq 结果表明 HSC 在细胞状态以及分化命运方向上具有异质性。为探究这背后的实质原因,我们需要追踪单细胞克隆的分化动态和 HSPC 的连续表达状态图谱。来自 Harvard 医学院和 Boston 儿童医院的研究者开发了一种可以捕获细胞状态的单细胞全转录组研究技术,通过研究不同时间点的细胞克隆命运,在数千个细胞中找寻单细胞状态与分化命运的内在关系。
细胞克隆状态与分化命运检测同步化
为同时捕获细胞的状态图谱与分化命运信息,作者设计了一套特殊的遗传编码来标记异质性祖细胞群,以便在分化过程中随时可以反复进行 scSeq。在经典的 lentiviral 病毒标记基础上,研究者改进了 DNA 编码(barcode)的标记方式,通过将 EF1α启动子连接 eGFP 蛋白序列,随后在 eGFP 序列 3′ UTR 端插入 28bp 随机片段,构建名为 Lineage and RNA recovery (LARRY) 的 DNA barcode。此编码文库库容可达~0.5 × 10^6,足以对 5000 cells 进行标记且 barcode 重复率小于 1%,再配合 scSeq 技术实现 LARRY 标记的表达检测。
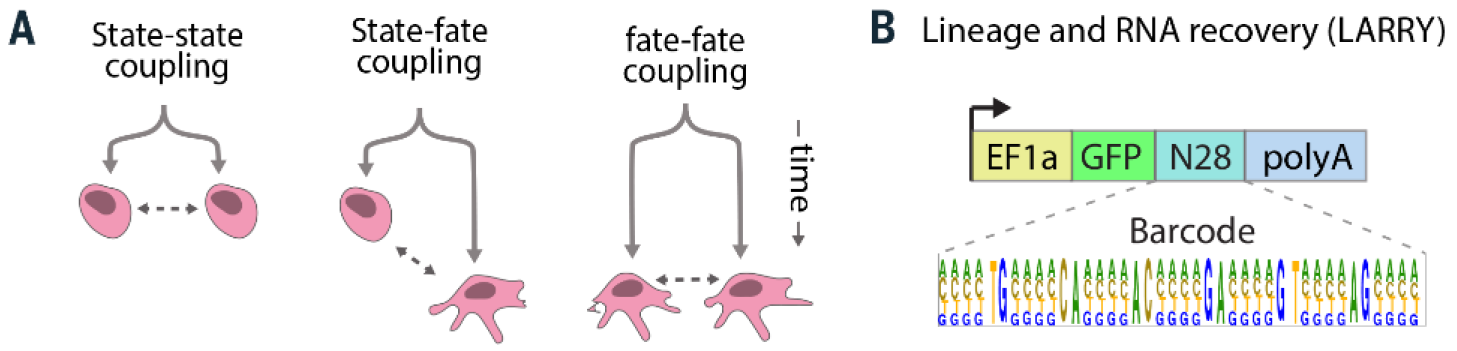
图 1:细胞状态图谱与分化命运图谱 coupling 示意(A);LARRY barcode 结构示意(B)
这一套 barcode 方案可以提供三类关键信息(图 1):分化初始阶段姊妹细胞的表达状态,初始阶段姊妹细胞与分化阶段子代细胞间的表达动态,以及不同分化方向终末细胞间的克隆联系。如果初代姊妹细胞(type 1)间在转录组学上表现相似,那么子代细胞的分化相近克隆(type 2)可以用来揭示单细胞分化过程中的基因表达动态。上述方案可在不需要分离或标记特定祖细胞群的基础上,呈现细胞群的连续分化表达的命运图谱。
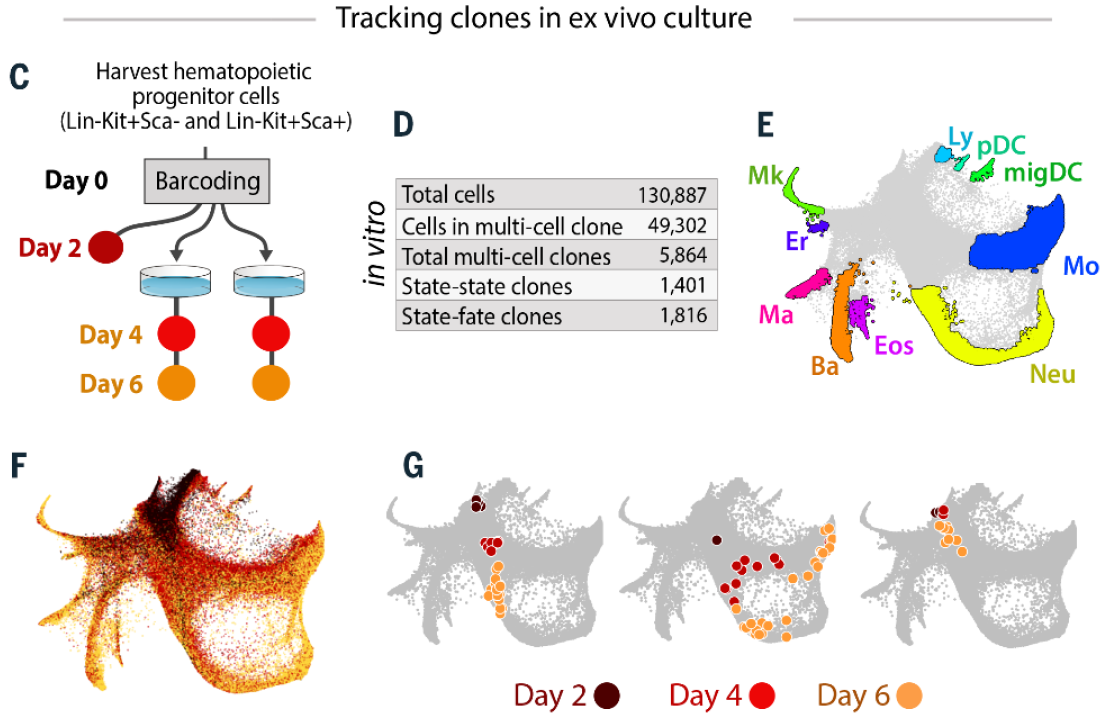
图 2:In vitro 组 HSPC 谱系追踪结果
为厘清 HSPC 的分化命运抉择,研究者使用 LARRY 标记方法,同时进行了体外培养(in vitro)和体内移植(cells transplanted in vivo)两种方案的研究。对于 in vitro 实验,作者使用 Sony SH800 自动化分选仪以极高的活性和纯度将 Lin-Sca-Kit+和 Lin-Sca1 +Kit+两类祖细胞分选出来,随即进入体外培养来探究多谱系分化动态。在转染 LARRY barcode 后细胞会经历两天的病毒整合和扩增,大约会发生三次增殖活动。在 day 2 取 50% 细胞进行 scSeq 记录细胞初始状态(early state),剩余细胞经过扩增后分别于 day 4(30% cells)和 day 6(remaining cells)再分别以 scSeq 记录细胞末期状态(late state)。In vivo 组则选用 Lin-Sca(hi)Kit+祖细胞,转染 LARRY barcode 后整合病毒表达和扩增。在 day 2 取 40% 细胞进行 scSeq 记录 early state,剩余细胞移植到 10 只辐照小鼠体内,经过一至两周循环扩增后再收集样本进行 scSeq 记录细胞 late state。

图 3:In vivo 组 HSPC 谱系追踪结果
在两套实验方案中,研究者分别收集到了 130,887(in vitro)和 182,173(in vivo)scSeq 转录组信息,共统计到 5864 clones 和 7751 clones,其中约有 38% 和 63% 的细胞属于同一种 clone,而在整个分化的 early state 和 late state 时间点中存在连续动态变化的 clones 各自有 1816 和 817。
通过 SPRING plots 将上述单细胞转录组数据可视化分析,in vitro 组 progenitor 呈现出从多能祖细胞(multipotent progenitors, MPPs)到九类成熟细胞的连续分化状态图谱(图 2E 和图 3J):erythrocytes (Er), megakaryocytes (Mk), basophils (Ba), mast cells (Ma), eosinophils (Eos), neutrophils (Neu), monocytes (Mo), dendritic cells (plasmocytoid pDC; Ccr7 + migratory migDC) 和 lymphoid precursors (Ly)。在图示的细胞状态图谱中,细胞分化轨迹表现出了显著的连续分化行为,包括单谱系分化、多谱系分化和祖细胞的自我更新(图 2 G)。In vivo 组 progenitor 也表现出同样的分化行为,从 MPPs 到多种状态的 neutrophil 成熟,以及 DCs, Mo, Er, B, T 和 Ba cells 群体的成熟。通过这两套方案,课题组成功地实现了单细胞状态图谱和克隆谱系分化命运的同时表征。
克隆分化动态鉴定早期细胞转录命运界限
有了 LARRY 标记方法的帮助,通过分析每个克隆在多个时间点上的 scSeq 数据即可实现单个细胞的谱系分化动态表征。但需要注意的是,这种方法的准确性极度依赖于初始时间点姊妹细胞间的相似性。如图 4 所示,在 day 2 初始姊妹细胞状态中,约 70% 姊妹细胞属于相同或邻近簇群。尽管存在约 10% 的偏离姊妹细胞群, 使得细胞分化命运界限的分辨率有所降低,但我们仍可通过克隆轨迹分析,成功推断出单细胞分化轨迹。
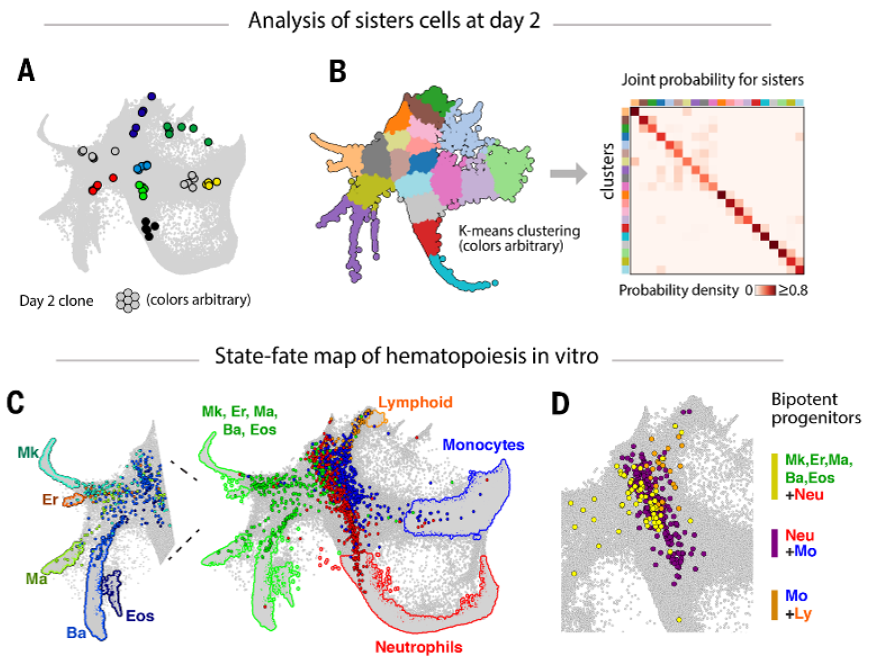
图 4:Early state 细胞转录表达状态与分化命运界限分析
在 in vitro 组数据中,研究者记录了所有 day 2 时间点的细胞状态和命运图谱。单一谱系克隆的 SPRING 图显示了良好的命运抉择,在祖细胞分化后的不同成熟细胞的重叠处,可见明显的 bi-potent 克隆对于命运抉择的界限位置,表明了祖细胞群并未选择离散的分化方式,取而代之的是结构性的连续分化轨迹。此外 bi-potent 克隆可形成额外的命运分支,说明细胞分化程序可在某些情况下独立于命运抉择程序之外自主发生。
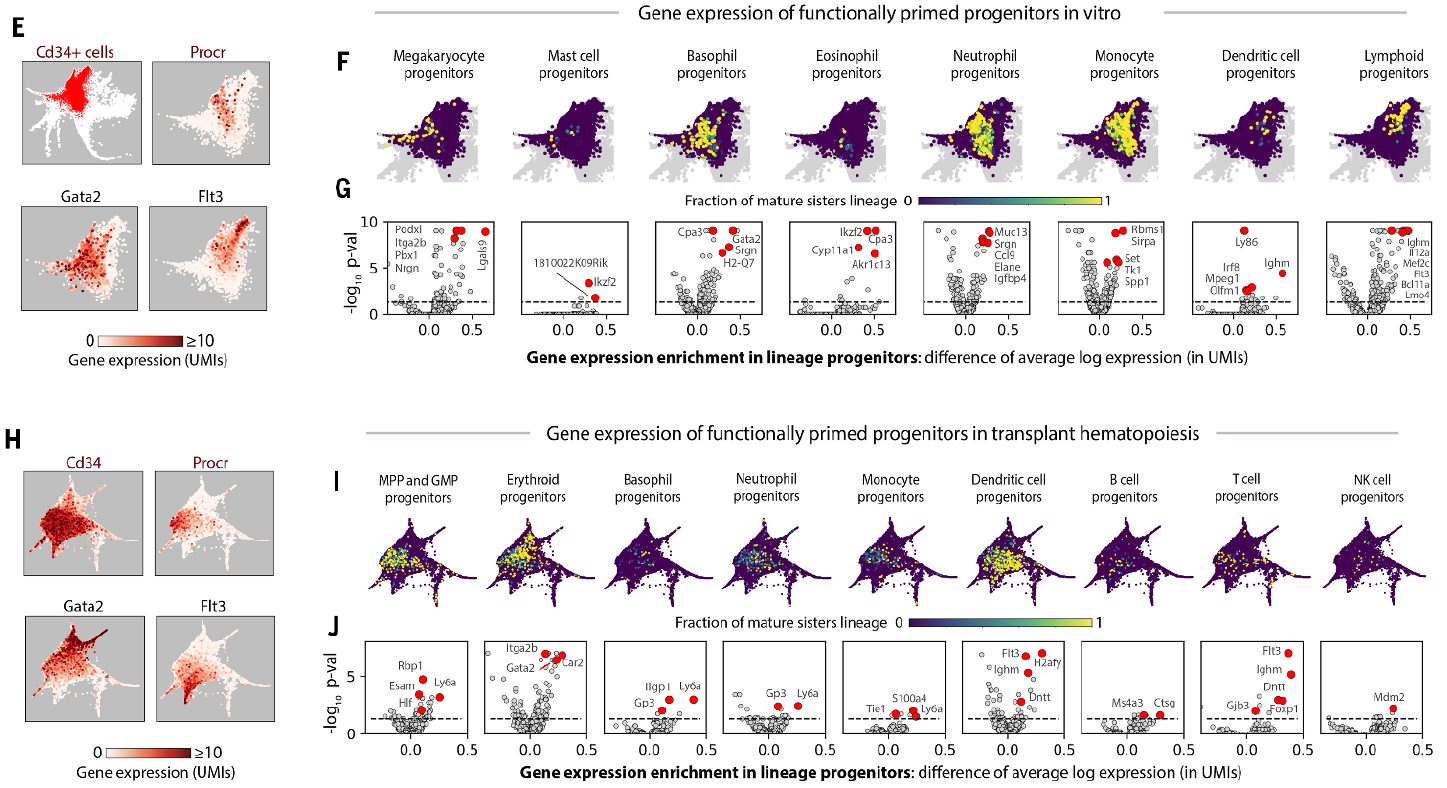
图 5:In vitro 组以及 in vivo 组祖细胞分化谱系追踪
通过基因表达的异质性分析,研究者确定了上述连续分化群体的亚群分布和可能的终末分化命运。处于 day 2 的早期多能祖细胞 CD34 +主要表现为干细胞 marker(Procr)表达,两侧分别为表达 Gata2 的红系细胞群和表达 Flt3 的淋巴样初始细胞(图 5E)。通过克隆叠加,可以看到各功能特异性谱系亚群与 CD34 +祖细胞的分化图谱严格一致,Mk,Ba,Ma 和 Eos 亚群均来源于 Gata2 +一侧;每个亚群的分化基因表达图谱也确认了造血过程中所有已知的分化 marker 以及潜在的未知 marker,如髓系白血病 marker(Ikzf2)高表达于 Ma 和 Eos 亚群,而少见于 Mk 和 Ba 亚群。In vivo 组的研究结果同样支持上述结论,尽管终末状态的亚群细胞相对较少,但分化命运均可与相应的选择 marker 一一对应。
总结来说,功能发育谱系与祖细胞的分化连续状态息息相关,并伴随着特定基因的表达变化而相应地改变如转录因子在内的功能状态表达。
scSeq 表达图谱能否有效预测细胞分化命运?
影响细胞分化命运的因素复杂多变,包括细胞与外在环境的接触、染色质状态、相关基因表达和随机的分子作用等。scSeq 技术仅可提供有限的细胞状态表达信息,我们无法以此构建完整无缺的状态图谱,那么随之而来的问题是 scSeq 数据究竟可以在多大程度上预测细胞分化命运呢?
为评估这一可能性,作者参考了两种机器学习算法(logistic regression & neural network)。以现有的 scSeq 数据为基础,通过对随机基因簇、转录因子、分化驱动基因和高度动态变化的基因进行演算,结果发现转录因子组的预测结果仅比随机基因簇组高~10%,而分化驱动基因组则可持续性地表现出差异性(图 6 A/B),说明 HSP 群体基因表达图谱的预测内容可有效覆盖数百个分化驱动基因,以及部分转录因子。
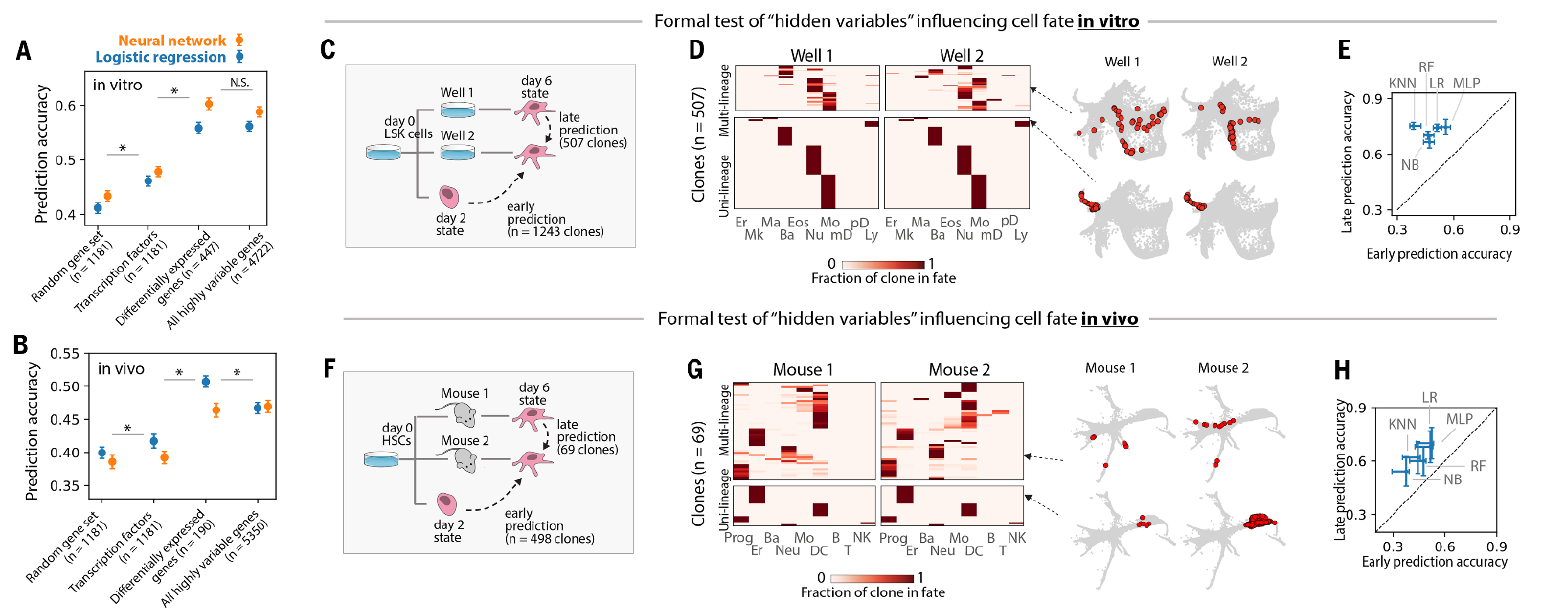
图 6:scSeq 数据预测细胞分化命运
细胞分化命运预测的准确性会受多种随机因素影响,这其中也包括了 scSeq 数据未能捕获的某些信息。如果这种隐藏变量真的存在,那么 scSeq 数据分析归类细胞亚群功能的准确性将大打折扣。通过 early state 和 late state 的细胞命运预测,课题组尝试验证上述变量是否真实存在。如果不存在,相邻姊妹细胞间的共同状态信息将随着分化而逐渐消失;如果存在,那么影响细胞分化的隐藏变量将会使姊妹细胞间的突变信息逐渐增多。
为比较哪个阶段的状态信息可更准确地预测分化命运,研究者对 early state 姊妹细胞和 late state 细胞表达状态进行了机器学习算法分析。演算结果发现,early state 信息可以为细胞分化命运的预测提供 60% 和 52% 的准确度(图 6 E/H),而 late state 信息可以预测提供 76% (in vitro) 和 70% (in vivo) 的准确度。因此 late state 更能反映姊妹细胞间分化命运的预测准确性,说明在细胞状态图谱中仍然存在 scSeq 技术无法捕获的隐藏变量,这些变量可以导致分化命运的不同抉择,即 scSeq 技术仍不足以准确描绘功能区分明确的祖细胞状态图谱。
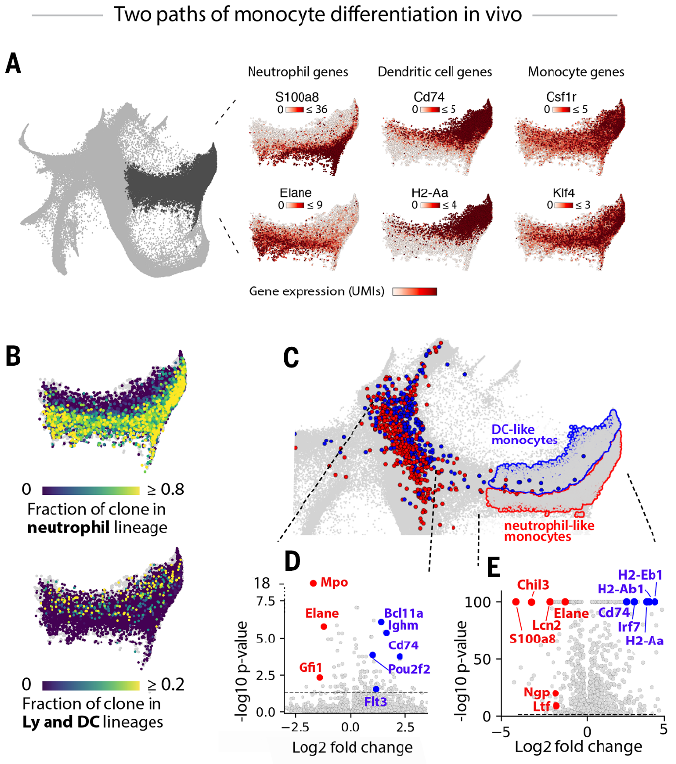
图 7:Monocytes 分化路径的谱系分析
克隆谱系分析,可以提供更多的 scSeq 数据无法揭示的分化路径信息。在前面的 dataset 中,我们可以看到 monocytes 的分化连续谱系:从 neutrophil-like type 到 DC-like type(图 7 A/C)。但在克隆相关性上,monocytes 与 neutrophils 以及 DCs 均没有显著性的紧密联系,故而 monocytes 在分化表型轨迹上与其他谱系的克隆间具有明显的独特性。作者分别检测了 day 2 和 day 6 姊妹细胞的 DC/neutrophil-like 亚群,基因表达图谱显示, neutrophil-like 以及 DC-like 亚群细胞在基因表达上表现出明显不同,这种迥异的分化轨迹可能对应着 MDP 和 GMP 亚群的分化路径。
造血作用中祖细胞分化命运的预测准则
通过前述的大量实验数据分析,作者整理出细胞分化轨迹的三类预测准则:
1. scSeq 数据模型不足以 100% 预测分化命运
研究者使用了三种预测模型:Population Balance Analysis (PBA), WaddingtonOT (WOT) 和 FateID,以 day 2 状态为起点,依据转录组数据预测 day 6 克隆状态(图 8)。三种模型均可与实际分化结果保持较好的一致性,但 CD34 +克隆轨迹分析显示 monocyte 与 neutrophil 存在分化交叉点,而这是预测算法未能有效推算的信息。预测模型使用分化调控基因的表达图谱进行演算,但 scSeq 数据有时可能无法全部捕获并呈现某些潜在的分化调控基因。因此在缺少谱系信息的情况下,预测算法可能会得出分化抉择演算错误的结果。

图 8:细胞分化命运图谱
2. Pseudotime 分析捕获细胞分化轨迹
常规中的 scSeq 数据分析中,一个重要目标是通过定义 pseudotime 时间流将分化动态中的基因表达依次排列开来,以此构建分化轨迹。到目前为止,我们仍旧还不清楚单细胞动态分化中的具体速率,以及其是否可以逆转分化动态。
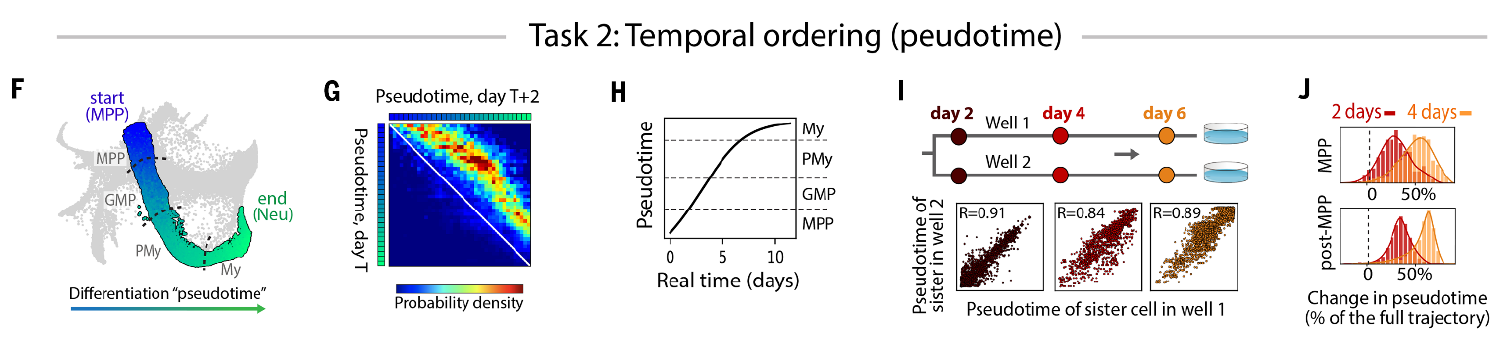
图 9:Pseudotime 分析描绘细胞分化轨迹
在 neutrophil 亚群分化过程中,研究者测试了 pseudotime 分析方法究竟可以多大程度地准确描述细胞亚群的分化动态轨迹。通过对 MPPs,GMPs,promyelocytes (PMy) 和 myelocytes (My) 多个样本亚群连续两天的克隆分析,结果发现在 pseudotime 时间流上表现出恒定向前的分化速率(图 9),例如 MPP 细胞分化至 myelocyte 需要 10 days,这与以往的研究结果一致;同时对姊妹细胞不同时间点的分化 pseudotime 流逐步分析,发现 day 2 与 day 6 节点细胞的扩增分化轨迹也保持了一致的步调。因此毫无疑问地, pseudotime 分析方法可用于描绘细胞发育的分化命运图谱。
3. 细胞状态图谱与分化谱系层级的协同一致
当细胞在分化轨迹中面临多方向选择时,基于细胞转录图谱与克隆谱系的高度一致性,人们选择使用 scSeq 技术来预测细胞分化谱系。但如前所述,上述预测模型并未考虑到下面两点因素:1)非同种分化克隆可能存在相似的终末细胞状态;2)即便来自于同样的祖细胞系,由于非对称分布,分化成熟的细胞群也可能保持不同终末状态。
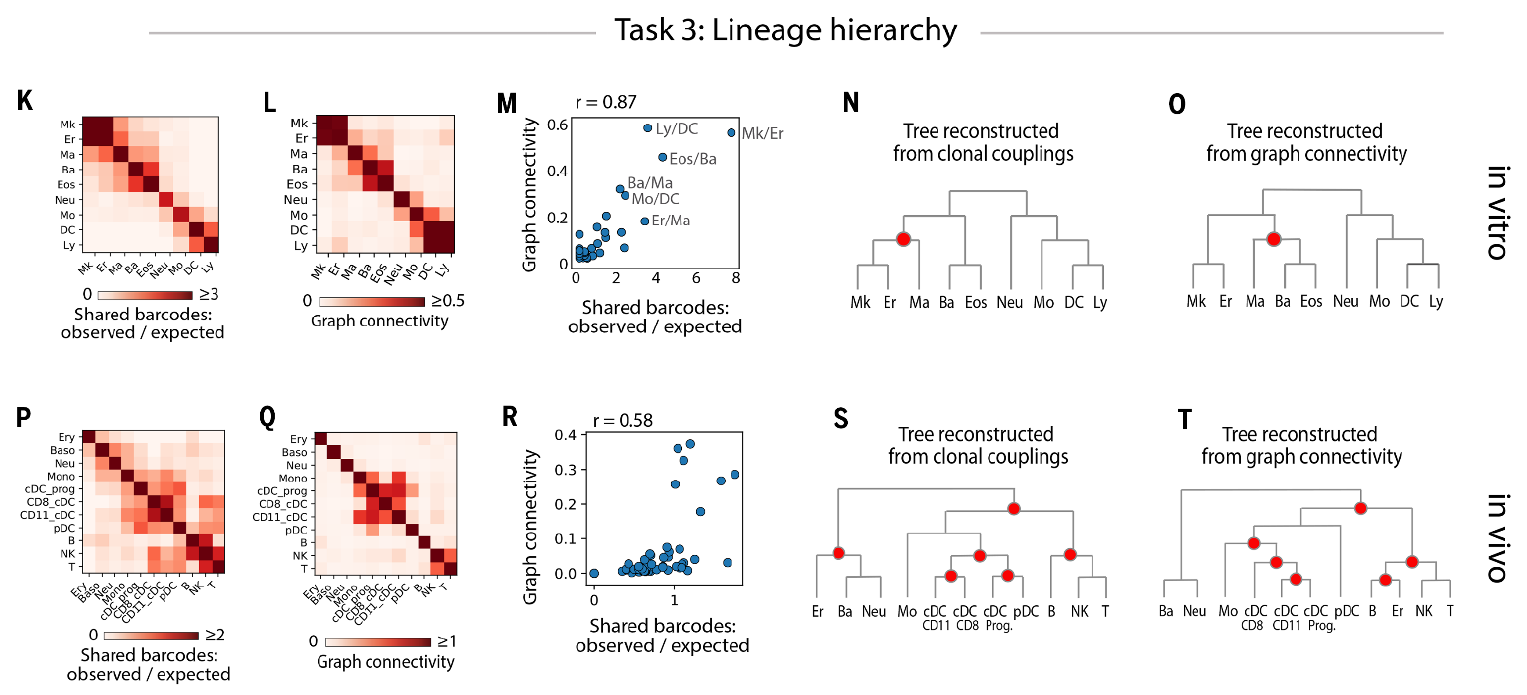
图 10:细胞状态图谱与分化命运图谱的联合演算
为使用谱系克隆和转录状态图谱构建真实准确的分化命运层级,研究者们系统比较了每对分化姊妹细胞的状态相似性和和克隆分化数目(图 10)。在 in vitro 组中当分别用状态图谱和克隆谱系构建分化路径后,结果显示两者的分化路径基本一致,仅在 mast cells 的分化节点上有所区别;而在 in vivo 组中,状态图谱与克隆谱系间的协同一致性明显降低,这可能与 in vivo 组复杂的分化环境以及更长的分化时间有密切联系。
Weinreb 等人开发的 LARRY barcoding 文库使得研究者们可以实现精确的谱系追踪。与基于 CRISPR 编辑的谱系追踪手段相比, LARRY 标记手段的不同点在于其不再仅将细胞的状态图谱作为终末变量,而是结合时间流实现拟时分析。相比于 CRISPR 的复杂操作,LARRY 更为简单易于实现,不需要建立谱系发育树;具有更高的单细胞 barcode 覆盖率;同时也不需要对细胞进行过多编辑。但 LARRY 标记手段也有其缺陷,就目前的技术而言 LARRY 无法描绘一个细胞周期内的细胞状态图谱,但 LARRY 仍可以无偏差的方式实现细胞的早期基因表达图谱与分化命运抉择间的深度联系。
通过 LARRY 标记手段,人们可以在 scSeq 数据的基础上,实现对不同组织样本的细胞状态图谱和分化命运图谱的描绘,并结合两者来帮助研究者们深入理解干细胞的发育分化行为机制。
Reference:
Weinreb et al., Science 10.1126 /science.aaw3381 (2020)









